核糖核苷酸还原酶
| A+医学百科 >> 核糖核苷酸还原酶 |
| ribonucleoside-diphosphate reductase | |||||||
|---|---|---|---|---|---|---|---|
|
|||||||
| 识别码 | |||||||
| EC编号 | 1.17.4.1 | ||||||
| CAS号 | 9047-64-7sy | ||||||
| 数据库 | |||||||
| IntEnz | IntEnz浏览 | ||||||
| BRENDA | BRENDA入口 | ||||||
| ExPASy | NiceZyme浏览 | ||||||
| KEGG | KEGG入口 | ||||||
| MetaCyc | 代谢路径 | ||||||
| PRIAM | 概述 | ||||||
| PDB | RCSB PDB PDBe PDBsum | ||||||
| 基因本体 | AmiGO / EGO | ||||||
|
|||||||
核糖核苷酸还原酶 (英文:Ribonucleotide reductase.(RNR), 别称为 核糖核苷二磷酸还原酶) 他是一个酵素,功能为把核苷酸催化为脱氧核糖核苷酸.[1] 脱氧核糖核苷酸常被使用于DNA的合成。而且在活体中物中,这个借由核糖核苷酸还原酶来催化的反应,受到非常严格的调控管理。[2] 此外,核糖核苷酸还原酶在调节DNA的合成的总速率中扮演一个非常关键的角色,可以让我们在细胞分裂和DNA 复制时,DNA和细胞质量维持在固定比率。[3]核糖核苷酸还原酶还有一个比较特别的调控方法,就是借由自由基机制来进行反应。[4][5]核糖核苷酸还原酶的基质有 ADP, GDP, CDP and UDP. dTDP (脱氧胸苷二磷酸)是从dTMP (脱氧胸苷一磷酸)而来,借由(胸苷激酶)催化而形成.
目录 |
结构
核糖核苷酸还原酶是一个铁离子依赖型的酵素,而且此酵素在DNA的合成中是一个必要的角色。第一型的核糖核苷酸还原酶是由一个比较大的RNR1和一个比较小的RNR2,这两个亚基所形成一个异二聚体四聚体。由于核糖核苷酸还原酶可以催化脱氧核糖核苷酸(dNTPs)的从头反应,脱氧核糖核苷酸是DNA合成的前驱物,在细胞增殖中是一个必要的一员。
在人体中,RNR1亚基是由RRM1基因来编码,同时他的异构型态RNR2是由RRM2基因和RRM2B基因来编码的:
- 1.含有220[[N-terminus|N-端]残基的主要螺旋结构。
- 2.包含480个残基,有十股α/β的结构。
- 3.包含70个残基比较小一点,有五股 α/β的结构。
在Pfam中,第二域名已被解释为两个单独的域:
- 1.一个比较小,全部由α组成的N端域。
- 2.一个比较大,全部由β组成的C端域。
RNR2含有一个亚铁中心和一个稳定的酪氨酰 自由基. 在Escherichia coli|大肠杆菌中,酪氨酰自由基位于Y12) 提供稳定的自由基给第一型的RNR2亚基。[7] 在 Aedes aegypti|埃及斑蚊体中, 酪氨酰自由基位于位置Y184。[8] 酪氨酰自由基通常被深深的埋在蛋白质的疏水环境中,而这个地方通常是在常使用稳定酪氨酰自由基的铁离子中心旁边。 2μ-氧联铁杆的结构是由铁结合位点的配体所提供的: 有4个胺基酸[天冬氨酸 (D146), 谷氨酸 (E177, E240, and E274)] 和两个组氨酸s (H180 and H277).[8] 发生在RNR2的C端和RNR1的C端的关联之间。[6] 酵素的活性依赖于RNR1和RNR2这两个基质的关联性。 活化中心包含从亚铁中心(RN1R)来的活化双硫醇基团和从RNR2亚基来的酪氨酰自由基。
RNR2的其他残基, 如天冬氨酸(D273), 色氨酸 (W48),和和酪氨酸(Y356) 可以进一步稳定活化中心的酪氨酰自由基,来进行我们的电子转移。[6] 这些残基有助于从酪氨酸RNR2(Y122)转移字由基给半胱氨酸RNR1(C439)。电子的转移从RNR2酪氨酸(Y122)开始,而且继续从RNR2到色氨酸(W48),这些都是借由2.5奈米的RNR1酪氨酸(Y731)分离而来的。在活化位中,电子由RNR1转移到RNR2且经过酪氨酸(Y356 to Y731)然后再透过酪氨酸(Y730)到半胱氨酸(C439)。[9] RNR一级结构的的定点突变指出我们上述的那些残基全部都有参雨我们把自由基传颂到活化位的长距离运输。[6]
在埃及斑蚊中, RNR1保留最关键的氨基酸残基,比如天门冬氨酸(D64)和缬氨酸(V292 or V284),这些胺基酸异位调节中都是非常重要的; 脯氨酸 (P210 and P610), 亮氨酸 (L453 and L473)和甲硫氨酸 (M603)残基都是座落在疏水部分的活化位;半胱氨酸(C225, C436 and C451)残基会在活化位中参与去除氢原子和转移自由基电子;半胱氨酸 (C225 and C436), 天冬酰胺 (N434)和谷氨酸 (E441)残基与核糖基质结合;酪氨酸(Y723 and Y743)残基决定自由基的转移;半胱氨酸(C838 and C841)残基在活化位被用来再生双硫醇基团。[8]
功能
核糖核苷酸还原酶(RNR)这个酵素从头催化dNTPs。[10]核糖核苷5'-二磷酸(NDPs)催化核糖5 - 磷酸2号碳的环原反应,形成2'-脱氧衍生物还原2'-脱氧核糖核苷5'-二磷酸(dNDPs)。而这个反应在我产生自由基的时候会启动。由单一的反应来,RNR需要硫氧还原蛋白的双硫醇基团所提供的电子。当烟酰胺腺嘌呤二核苷酸磷 (NADPH)提供两个氢原子来环原硫氧还原蛋白的二硫化物基团时,我们的硫氧环原蛋白会进行再生。
三个类型的RNR都有非常相像的机制来还原NDPs,三者的不同处只在于产生自由基的部分。金属蛋白的结构中含有特定的金属离子和电子提供者,所有类型都是使用自由基化学。[6] 第一型的还原酶使用亚铁离子中心和三价铁转化为游离的酪氨酰自由基,通常还原NDP基质都要在有氧的情况下进行。第一型的还原酵素贝分为IA和IB两类型,分配管制不同的调节作用。IA型还原酶通常分布在真核生物,真细菌, 噬菌体,和病毒中.而IB型的还原酶则被发现在真细菌中。 IB型还原酶也可以借由双核锰中心稳定的产生自由基。
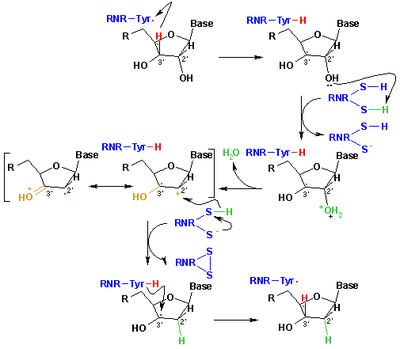
还原反应的催化机制
核糖核苷酸变成脱氧核糖核苷酸的这个还原机制就是我们的下图。第一步骤我们先把3'-H的基质1'通过自由基Cys439。然后借由Cys225和Glu441的催化把核糖核苷酸的2号碳位子消除一个水分子。第三个步骤在之前的质子从Cys462转移到Cys225后,氢原子从Cys225转移到2'-羰自由基3的2号碳位子。在最后一个步骤中,获得阴离子的自由基双硫桥和和闭壳层酮中间体4。在几个2'-取代基的类似物的转化过程中这个中间物被确定,以及和酶的突变体作用过的天然基质。[11] 可以借由Cerqueira 等人的研究来了一些使用整个R1蛋白的机制.[12][13]
调节
第一型RNR包含了RNR1和RNR2两个亚基,和形成异源二聚体的四聚体有关。[4] RNR1包含变构点,还有调节基质特异性和活性的功能。 4个核糖核苷酸的期中一个接合上活性位,取决于变构的配制。
RNR的调节是为了维持dNTPs的量。当ATP结合上异构活化位时会活化RNR。相反的,当dATP结合到异构活化位时,RNR会失去活性。[6]除了控制活性外,这种异位调节机制,还可以调节基质的专一性还有确保酵素所制造的dNTP是等量的。[6]所有的种类在异构为上结合ATP或dATP都会诱导胞苷5'-二磷酸(CDP)和尿苷5'-二磷酸(UDP)的还原;2'-脱氧鸟苷-5'-三(三磷酸)诱导还原腺苷5'-二磷酸(ADP); 2'-脱氧胸苷5'-三磷酸(dTTP的)诱导还原5'-鸟苷二磷酸(GDP)。
有趣的是,IB型的还原酶不会被dATP抑制因为他们在异构活化位上缺乏了大概50个N端的胺基酸。[14]真核细胞的IA型还原酶有一个附调控机制,就是当dNTPs浓度过高时,他会关闭她的合成机制。.这个机制可以保护我们的细胞不会因为dNTP的过剩造成的细胞毒性或突变效应而造成细胞死亡或DNA的损害。[15][16]
RNR 可以使用异位调节的morpheein模型.[17]
RNR1和RNR2抑制剂
通常类型I的RNR抑制剂可以被分成三种:转译抑制剂,可以中止酵素的合成。二聚化抑制剂,可以预防两个RNR亚基的关联(R1和R2)。最后是催化抑制剂可以让R1亚基或R2亚基失活。.[12]
第一型的RNR和第二型的RNR的C端很像,都可以被肽类抑制。
参考资料
- ↑ Elledge SJ, Zhou Z, Allen JB. Ribonucleotide reductase: regulation, regulation, regulation. Trends Biochem. Sci.. March 1992, 17 (3): 119–23. doi:10.1016/0968-0004(92)90249-9. PMID 1412696.
- ↑ Torrents E, Aloy P, Gibert I, Rodríguez-Trelles F. Ribonucleotide reductases: divergent evolution of an ancient enzyme. J. Mol. Evol.. August 2002, 55 (2): 138–52. doi:10.1007/s00239-002-2311-7. PMID 12107591.
- ↑ Herrick J, Sclavi B. Ribonucleotide reductase and the regulation of DNA replication: an old story and an ancient heritage. Mol. Microbiol.. January 2007 pmid = 17229208, 63 (1): 22–34. doi:10.1111/j.1365-2958.2006.05493.x.
- ↑ 4.0 4.1 Eklund H, Eriksson M, Uhlin U, Nordlund P, Logan D. Ribonucleotide reductase--structural studies of a radical enzyme. Biol. Chem.. August 1997, 378 (8): 821–5. PMID 9377477.
- ↑ Stubbe J, Riggs-Gelasco P. Harnessing free radicals: formation and function of the tyrosyl radical in ribonucleotide reductase. Trends Biochem. Sci.. November 1998, 23 (11): 438–43. doi:10.1016/S0968-0004(98)01296-1. PMID 9852763.
- ↑ 6.0 6.1 6.2 6.3 6.4 6.5 6.6 Jordan A, Reichard P. Ribonucleotide reductases. Annu. Rev. Biochem.. 1998, 67 (1): 71–98. doi:10.1146/annurev.biochem.67.1.71. PMID 9759483.
- ↑ Högbom M, Andersson ME, Nordlund P. Crystal structures of oxidized dinuclear manganese centres in Mn-substituted class I ribonucleotide reductase from Escherichia coli: carboxylate shifts with implications for O2 activation and radical generation. J. Biol. Inorg. Chem.. March 2001, 6 (3): 315–23. doi:10.1007/s007750000205. PMID 11315567.
- ↑ 8.0 8.1 8.2 Pham DQ, Blachuta BJ, Nichol H, Winzerling JJ. Ribonucleotide reductase subunits from the yellow fever mosquito, Aedes aegypti: cloning and expression. Insect Biochem. Mol. Biol.. September 2002, 32 (9): 1037–44. doi:10.1016/S0965-1748(02)00041-3. PMID 12213240.
- ↑ Chang MC, Yee CS, Stubbe J, Nocera DG. [http//www.ncbi.nlm.nih.gov/pmc/articles/PMC406436/ Turning on ribonucleotide reductase by light-initiated amino acid radical generation]. Proc. Natl. Acad. Sci. U.S.A.. May 2004, 101 (18): 6882–7. doi:10.1073/pnas.0401718101. PMID 15123822. PMC 406436.
- ↑ Cox, Michael; Nelson, David R.. Lehninger Principles of Biochemistry. San Francisco: W. H. Freeman. 2008. ISBN 0-7167-7108-X.
- ↑ Cerqueira NM, Fernandes PA, Eriksson LA, Ramos MJ. Ribonucleotide activation by enzyme ribonucleotide reductase: understanding the role of the enzyme. J Comput Chem. December 2004, 25 (16): 2031–7. doi:10.1002/jcc.20127. PMID 15481089.
- ↑ 12.0 12.1 Cerqueira NM, Pereira S, Fernandes PA, Ramos MJ. Overview of ribonucleotide reductase inhibitors: an appealing target in anti-tumour therapy. Curr. Med. Chem.. 2005, 12 (11): 1283–94. doi:10.2174/0929867054020981. PMID 15974997.
- ↑ Cerqueira NM, Fernandes PA, Eriksson LA, Ramos MJ. [http//www.ncbi.nlm.nih.gov/pmc/articles/PMC1386789/ Dehydration of ribonucleotides catalyzed by ribonucleotide reductase: the role of the enzyme]. Biophys. J.. March 2006, 90 (6): 2109–19. doi:10.1529/biophysj.104.054627. PMID 16361339. PMC 1386789.
- ↑ Eliasson R, Pontis E, Jordan A, Reichard P. Allosteric regulation of the third ribonucleotide reductase (NrdEF enzyme) from enterobacteriaceae. J. Biol. Chem.. October 1996, 271 (43): 26582–7. doi:10.1074/jbc.271.43.26582. PMID 8900130.
- ↑ Kunz BA. Mutagenesis and deoxyribonucleotide pool imbalance. Mutat. Res.. 1988, 200 (1-2): 133–47. doi:10.1016/0027-5107(88)90076-0. PMID 3292903.
- ↑ Meuth M. The molecular basis of mutations induced by deoxyribonucleoside triphosphate pool imbalances in mammalian cells. Exp. Cell Res.. April 1989, 181 (2): 305–16. doi:10.1016/0014-4827(89)90090-6. PMID 2647496.
- ↑ T. Selwood and E. K. Jaffe.. Dynamic dissociating homo-oligomers and the control of protein function.. Arch. Biochem. Biophys.. 2011, 519 (2): 131–43. doi:10.1016/j.abb.2011.11.020. PMID 22182754. PMC 3298769.
外部连结
|
||||||||||||||||||||||||||
参考来源
| 关于“核糖核苷酸还原酶”的留言: | |
|
目前暂无留言 | |
| 添加留言 | |